导读:
抗生素耐药性是一个日益严重的健康问题,准确评估细菌的表型和基因型特征对于理解抗生素耐药性至关重要。然而,目前的方法主要关注不同菌落的个体表型或基因型特征。近期,厦门大学嘉庚创新实验室张惠敏团队提出了一种结合了数字微流控和单细胞基因组测序的创新方法(Digital-GPA),用于快速、准确分析单个细菌的基因型和表型,以识别抗生素抗性机制,具有直接处理临床样本和无需细菌培养的优势。相关研究以“Metabolic Labeling and Digital Microfluidic Single-Cell Sequencing for Single Bacterial Genotypic-Phenotypic Analysis”为题目发表于期刊《Small》。
本文要点:
1、本研究开发了一种基于数字微流控的自动化单细菌全基因组测序方法(Digital-GPA),用于抗生素耐药菌株的基因型和表型分析。
2、该方法通过高效分离出经荧光D-氨基酸(FDAA)标记的抗生素耐药细菌并对其进行测序,产生高质量的单细胞扩增基因组(SAG),并且能够识别主要和次要突变,精确定位具有不同耐药机制的亚株。
3、Digital-GPA可直接处理临床样本,无需细菌培养即可对耐药病原体进行检测和测序,随后提供抗生素敏感性的基因图谱,为分析难培养或生长缓慢的细菌提供了新的途径。
4、该方法通过在单细胞分辨率下提供准确且全面的抗生素耐药性分子图谱,显著加快了分析过程,有助于更好地理解和应对抗生素耐药性问题。
在这项研究中使用FDAA(荧光D-氨基酸)标记意义重大,其原因主要有以下几点:
1、FDAA标记可快速识别抗生素耐药细菌。耐药细菌即使在高浓度抗生素的作用下也能持续发出强烈荧光,而易感细菌则荧光减弱。
2、通过FDAA标记,可在数字微流控芯片上精确分离和捕获单个抗生素耐药细菌细胞。这是后续单细胞基因组测序的关键步骤。
3、FDAA标记提供了一种可靠的方法来衡量单个细菌细胞的代谢状态和抗菌药物敏感性表型,这对于关联抗生素耐药性的基因型和表型特征非常重要。
总之,FDAA标记技术是Digital-GPA能够在单细胞水平上同时确定抗生素耐药细菌的表型和基因型特征的关键组成部分。
Digital-GPA技术在处理临床样本时如何确保单细胞基因组测序的准确性和重复性,尤其是在面对复杂微生物群落的情况下?
1、FDAA标记:使用荧光D-氨基酸(FDAA)标记快速识别和选择活跃的抗生素耐药细菌,这些细菌即使在高浓度抗生素下也能保持代谢活性,从而确保了目标细胞的选择性。
2、数字微流控芯片:通过数字微流控技术(DMF)精确操控和隔离单个细菌细胞,减少了样本间的交叉污染,提高了测序的准确性。
3、多重置换扩增(MDA):采用MDA方法在芯片上进行单细胞基因组的指数级扩增,这种方法在小体积反应中提高了扩增的均匀性和效率。
4、优化的染色和捕获效率:通过优化亲水区域的尺寸和细菌悬液的浓度,提高了单细胞捕获的效率,确保了测序样本的代表性。
5、全面的质量评估:通过综合考量一系列质量评估指标(映射率、基因组完整性、污染程度等),对测序数据进行了严格的质量控制,从而确保了研究结果的准确性和可信度。
6、综合基因型和表型分析:通过同时分析细菌的基因型和表型,Digital-GPA能够相互验证两者的一致性,提高了对抗生素耐药性分析的准确性。
7、临床样本验证:在临床尿液样本中的应用表明,Digital-GPA能够准确识别具有不同耐药机制的亚株,并通过与传统的抗生素敏感性测试(AST)方法比较,证实了其准确性。
使用数字微流控技术进行单细胞测序具有以下优势:
1、可以在纳升级别的微滴内进行细胞分离、裂解和DNA扩增等关键步骤,提高了反应效率和均一性,减少了扩增偏差。
2、将细菌细胞封装在微滴中有助于避免外源性细菌污染,提高测序结果的可靠性。
3、集成了所有关键反应步骤,避免了从管式反应转移到其他设备时可能导致的样品损失或污染。
4、微滴操作可精确控制单个细菌细胞的捕获和分离,为后续的单细胞基因组测序奠定基础。
5、与传统的管式反应相比,数字微流控芯片所需的样品量更少,只需0.3μL,有利于分析低浓度细菌样本。
总之,数字微流控技术在单细菌基因组测序中显示出集成性、微型化和精确操控等优势,有助于提高测序结果的质量和可靠性,为抗生素耐药性分析提供更准确的信息。
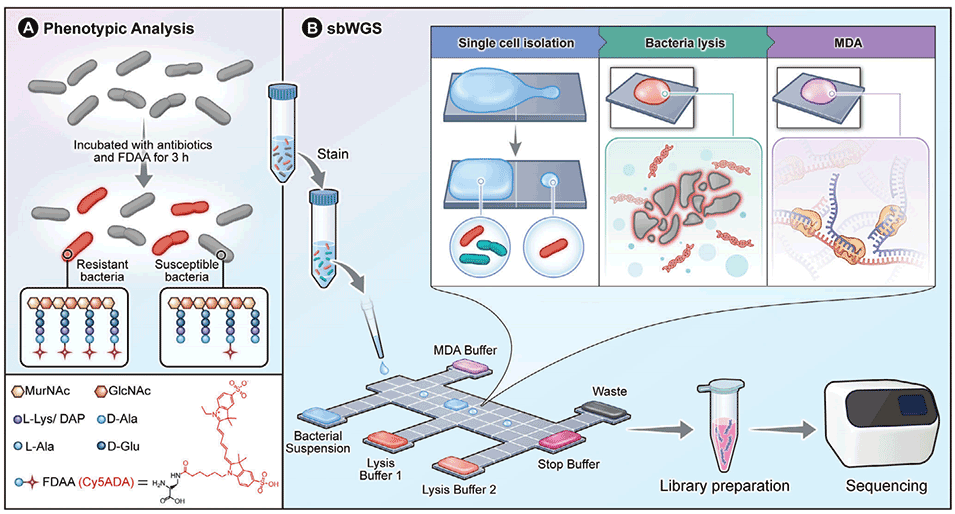
图1.单细菌基因型-表型抗生素耐药性分析的Digital-GPA工作流程示意图。
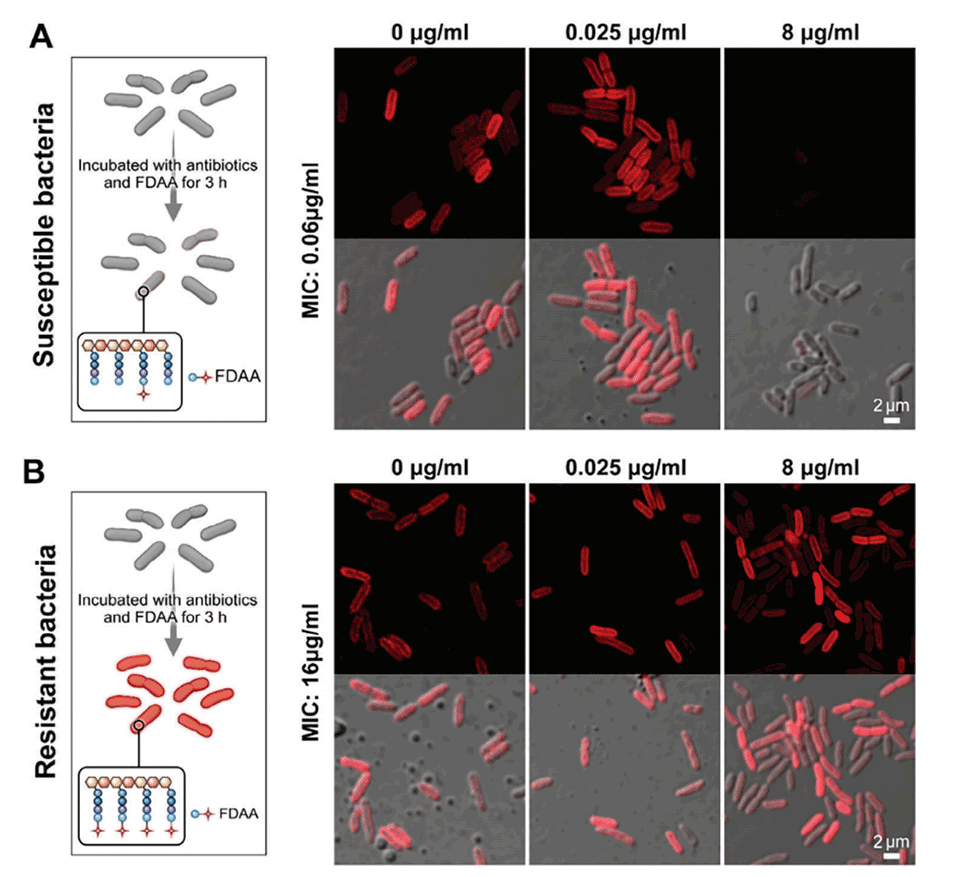
图2.不同左氧氟沙星(LEV)浓度下FDAA标记的敏感和耐药细菌的共聚焦荧光成像。
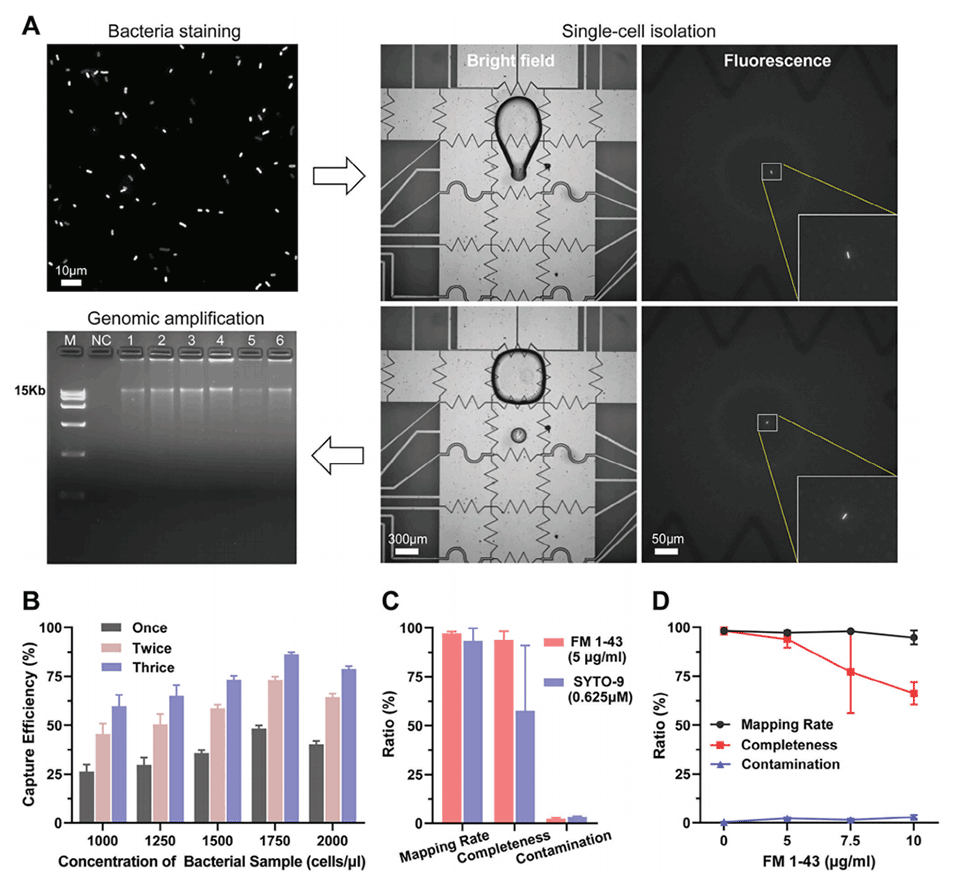
图3.数字微流控芯片上单细菌捕获和全基因组测序的表征。
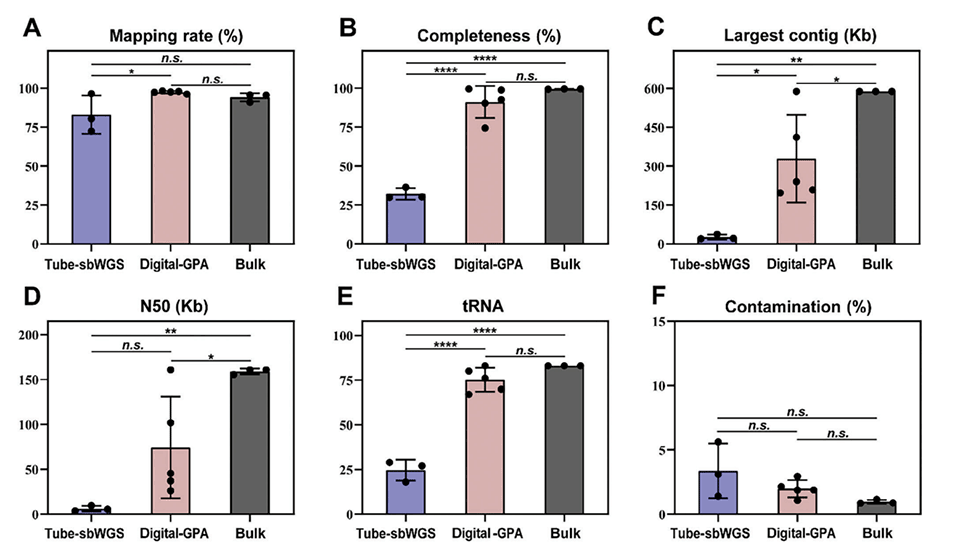
图4.Digital-GPA、Tube-sbWGS和批量数据之间的性能比较。
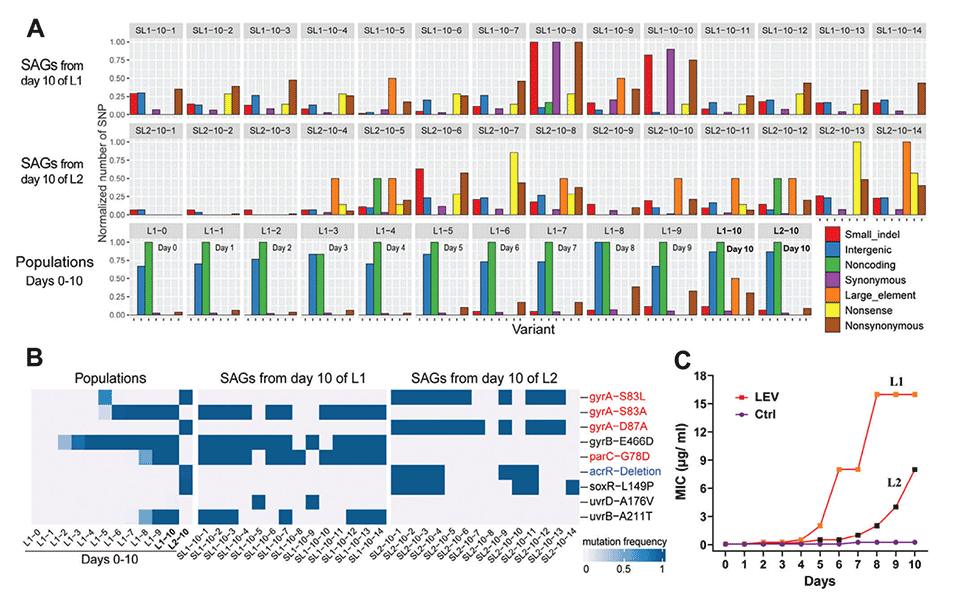
图5.LEV抗性进化实验中SAG与群体遗传变化的比较。
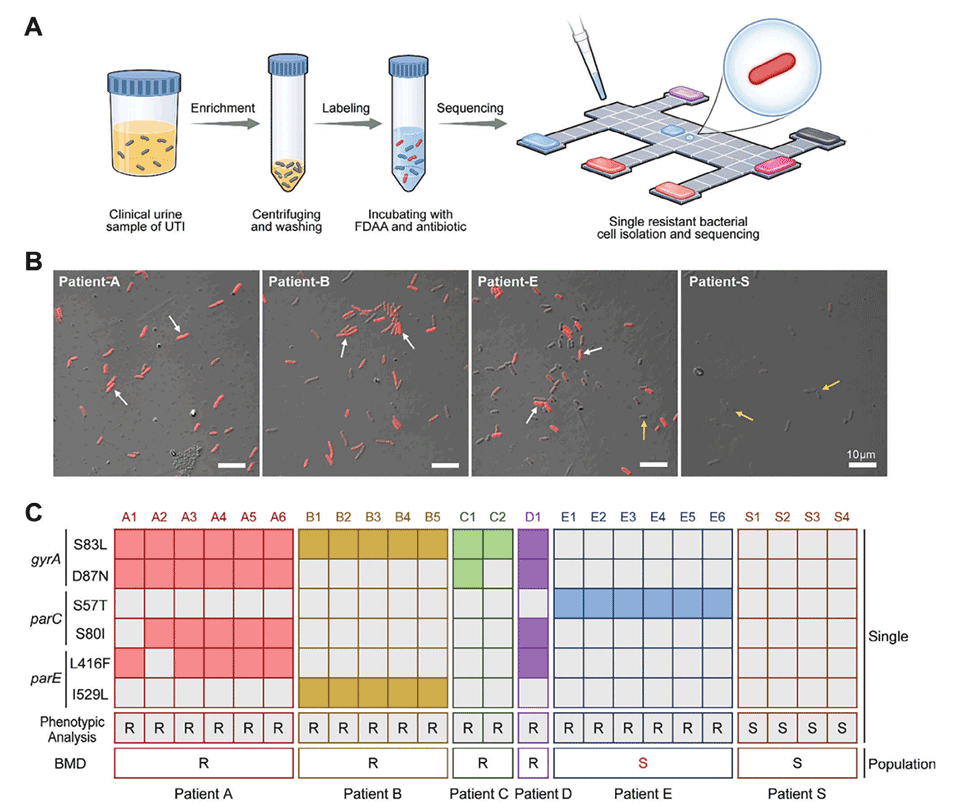
图6.Digital-GPA在尿路感染临床尿液样本中的应用。
原文链接:https://doi.org/10.1002/smll.202402177